5.3.2.1 - UPGMA (Sneath et Sokal 1973)
La méthode UPGMA [1] (Unweighted Paired Group Mean Arithmetic) permet de construire des arbres ultramétriques. Dans ces arbres, toutes les feuilles sont à équidistance de leur ancêtre commun. Cette propriété répond parfaitement au principe de l'horloge moléculaire qui suppose que les substitutions s'accumulent à une vitesse constante dans le temps et que les distances évolutives sont le reflet (strict) des temps géologiques qui sépare deux OTU de leur ancêtre commun.
Attention :
Si les distances évolutives ne répondent pas strictement au principe de l'horloge moléculaire, les arbres produits sont faux.
Méthode :
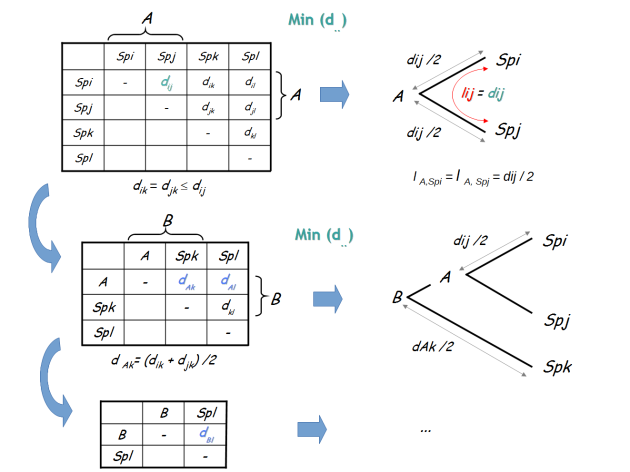
La méthode UPGMA consiste à regrouper récursivement[2] sous un même ancêtre les couples d'OTU présentant la distance évolutive la plus faible (fig. 5.41). La distance des OTU à cet ancêtre est égale à la moitié de la distance évolutive entre les OTU considérées.
Dans la matrice de distance, l'ancêtre inféré se substitue aux OTU qu'il regroupe. La distance de l'ancêtre aux autres OTU encore présentes dans la matrice est alors égale à la moyenne des distances entre les OTU en question et les OTU présentes aux feuilles sous l'ancêtre considéré
Ces étapes sont appliquées de manière répétées (récursivement) sur des matrices de plus en plus petites jusqu'à ce que l'ensemble des OTU soient inclus dans l'arbre final.
Interprétation
Dans l'arbre produit, chaque ancêtre est équidistant de toutes les feuilles du sous-arbre qu'il racine. Par construction on inclus les OTU depuis les plus semblables (qui ont un ancêtre commun récent) aux plus distants (ou divergent). Il est par conséquent orienté dans le temps. Il est raciné.
Si la méthode UPGMA est historiquement la première méthode proposée, on lui préfère la méthode du Neighbor-Joining, en particulier parce que cette méthode tolère des écarts à l'horloge moléculaire. En effet, au sens strict la méthode UPGMA est issue des méthodes de clusterisation et n'a de ce fait pas pour objectif de refléter des descendances évolutives, mais de regrouper des éléments (ici des OTU) sur la base d'une mesure de distance.
Exemple :
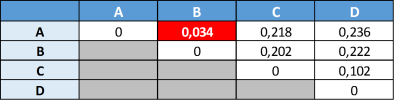
Construisons un arbre par la méthode UPGMA à partir de la matrice suivante obtenue en comparant des séquences orthologues dans quatre espèces différentes A, B, C et D :
La première étape consiste à repérer dans la matrice la valeur la plus faible, c'est à dire ici les deux espèces les plus proches. Il s'agit de 0,034, distance séparant A de B :
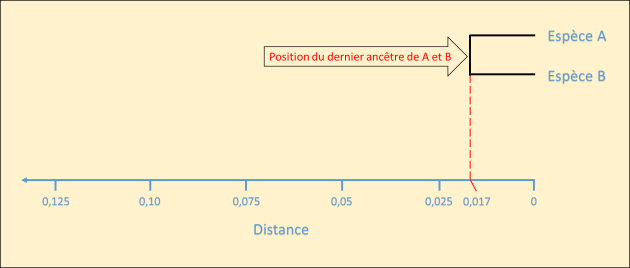
Il est alors possible de placer A et B sur l'arbre et de déterminer la position de leur dernier ancêtre commun à la moitié de la distance les séparant soit 0,017 (fig. 5.42).
La matrice initiale va donc pouvoir être simplifiée, A-B ne formant plus qu'une seule HTU. Les distances entre le groupe (A-B) et les autres OTU doivent être recalculées :
d(AB->C) = [d(A->C) +d(B->C)]/2 = (0,218+0,202)/2 =0,210
d(AB->D) = [d(A->D) +d(B->D)]/2 = (0,236+0,222)/2 =0,229
On obtient ainsi la matrice suivante :
Le principe se répète :
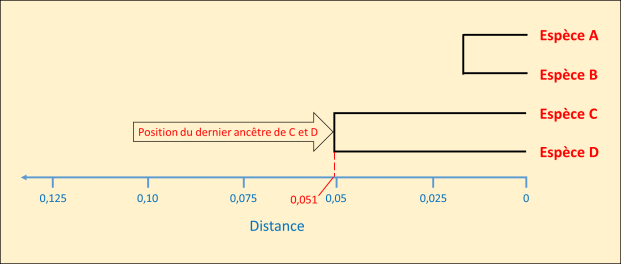
repérer la valeur la plus faible (0,102)
regrouper les OTU concernées (C et D),
calculer la position du dernier ancêtre commun (0,051)
compléter l'arbre (fig. 5.43).
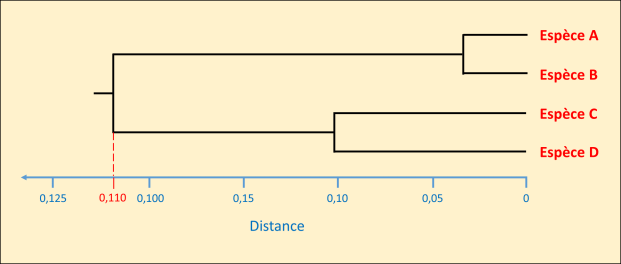
La dernière étape va consister à calculer la distance séparant le groupe A-B du groupe C-D :
d(AB->CD) = [d(A->C)+d(A->D)+d(B->C)+d(B->D)]/4 = [0,218+0,236+0,202+0,222]/4 = 0,219
Le dernier ancêtre commun aux groupes AB et CD peut donc être placé à la moitié de cette valeur soit 0,110 environ. L'arbre définitif obtenu est donné fig. 5.44.
Remarque :
Selon cet arbre obtenu, la distance entre A et C devrait être équivalente à celle entre A et D par exemple. On voit dans la matrice initiale que cela n'est pas totalement exact. La différence ici est faible mais il peut arriver que cette différence soit importante. Cela est dû au non respect de l'horloge moléculaire, ce que la méthode UPGMA ne prend pas en compte.