5.3.4 - Evaluation d'une phylogénie : le "bootstrap"
Les jeux de données utilisés dans les reconstructions phylogénétiques ne sont qu'un échantillon des caractères propres aux OTU considérés.
On peut se demander alors si les signaux évolutifs présents dans ces échantillons restituent de manière robuste/reproductible l'information que l'on aurait avec l'intégralité des caractères portés par ces OTU.
En d'autres termes, l'information restituée par l'échantillon peut-elle se généraliser à n'importe quel échantillon de même taille extrait de l'ensemble des caractères des spécimens étudiés ?
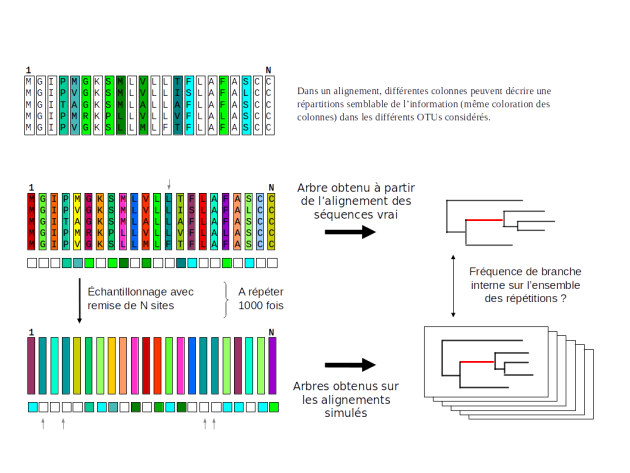
La méthode du bootstrap est une méthode statistique qui vise à répondre à cette question. Elle consiste à reconstruire un ensemble de données en pratiquant des tirages aléatoires avec remise parmi les caractères étudiés et à comparer les phylogénies obtenues.
Méthode :
Pour un ensemble de N caractères :
Tirage d'un caractère au hasard et remise
Répétition N fois du tirage : dans ce nouvel échantillon, certains caractères peuvent apparaître plusieurs fois alors que d'autres ne seront jamais tirés au sort (fig. 5.51).
Utilisation du nouvel ensemble pour faire un arbre
Recherche des branches communes avec l'arbre original.
Répétition un grand nombre de fois (100, 1000 fois) des étapes 1 à 4
Estimation de la fréquence à laquelle les branches de l'arbre initial sont retrouvées dans les arbres simulés.
La valeur obtenue, exprimée en pourcentage, est appelée bootstrap .
Une valeur de bootstrap forte décrit un signal fortement présent dans les données analysées, alors qu'un bootstrap faible traduit un signal peu marqué et donc peu fiable dans le jeu de données.
Attention : Le bootstrap n'est pas un indicateur de véracité
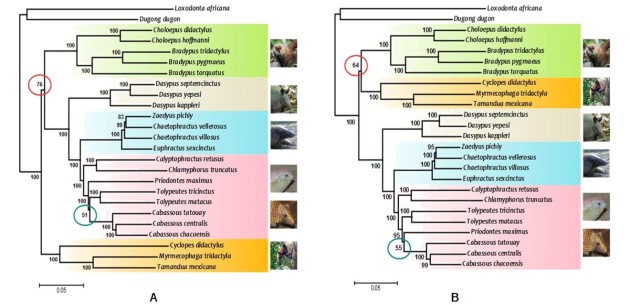
Les phylogénies A et B (fig. 5.52) ont été estimées à partir du jeu de données issu de la publication de Gibb et al. 2016[1] (ADN mitochondrial de différentes espèces de Xanarthres). L'arbre A a été inféré en appliquant le modèle d'évolution de Jukes et Cantor, alors que le modèle d'évolution HKY a été utilisé pour la construction de l'arbre B. Dans les deux cas 100 bootstraps ont été effectués.
On constate que les relations de parentés décrites à travers ces deux arbres sont globalement semblables et très fortement soutenues :
→ les valeurs de bootstrap (pour l'essentiel égales à 100) reflètent un signal évolutif fortement présent dans les données.
On peut cependant noter des incongruités dans le positionnement de certains taxons (noter les positions du groupe « orange » ainsi que la celle de Priodontes maximus) associées à des valeurs de bootstrap plus faibles. Ceci indique que le signal évolutif associé à ces groupes est moins présent dans le jeu de données analysées.
Si l'on s'attache à comparer les valeurs de bootstrap associées au positionnement du « groupe orange » par rapport au reste de l'arbre, on constate que la valeur de bootstrap dans l'arbre A (76) est supérieure à celle de l'arbre B (64). Il serait inapproprié d'interpréter cette valeur plus élevée comme un gage de plus grande véracité de la relation présentée dans l'arbre A par rapport à l'arbre B, car en effet c'est l'arbre B qui est le plus proche de la phylogénie de référence présentée dans l'article de Gibb[1]. Ainsi, le bootstrap ne précise pas le degré de vérité des relations évolutives décrites dans une phylogénie, il n'est que l'estimation de l'intensité du signal qui donne lieu au résultat observé.
Le bootstrap décrit donc la force d'un signal dans un jeu de données, mais ne décrit en aucune manière la véracité de ce signal. Ainsi, un signal peut être à la fois très fort et très faux.