Les complexes régulateurs
L'unité régulatrice PA 700 a quatre rôles :
elle fixe le substrat (récepteur),
elle facilite le dépliage et le passage de la chaîne polypeptidique vers l'intérieur du protéasome, par son activité ATPasique,
elle enlève les ubiquitines (qui sont recyclées) et finalement
elle active le protéasome par ouverture du pore formé par les sept sous-unités \(\alpha\) (voir figure 24).
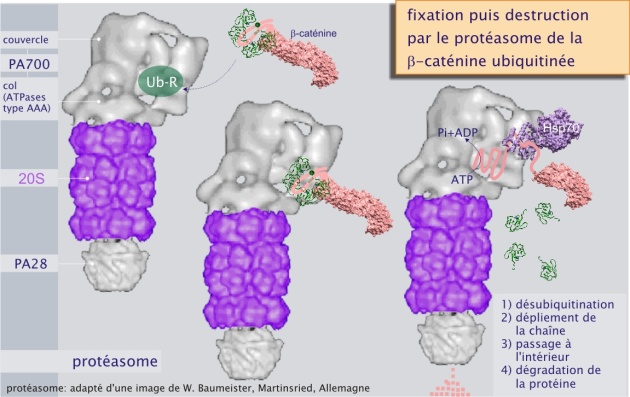
Ainsi la fixation d'une unité régulatrice au complexe 20 S permet l'accès à la chambre catalytique () et ceci est également vrai pour PA 28. C'est la raison pour laquelle elles sont aussi appelées « Proteasome Activator » (PA 28 ou PA 700). Par ce mécanisme, la cellule est capable de limiter la dégradation protéique non sélective. PA 28, beaucoup moins courant que PA 700, ne fixe pas les protéines ubiquitinées et son rôle reste donc à définir.
PA 700 comprend 20 composants au minimum, appelés Rpn's et Rpt's. Il est divisé en deux parties :
a/ la base qui fixe la particule 20 s et qui est constituée d'ATPases type AAA (Rpt1-6) et de non-ATPases (Rpn1,2 et 3) et
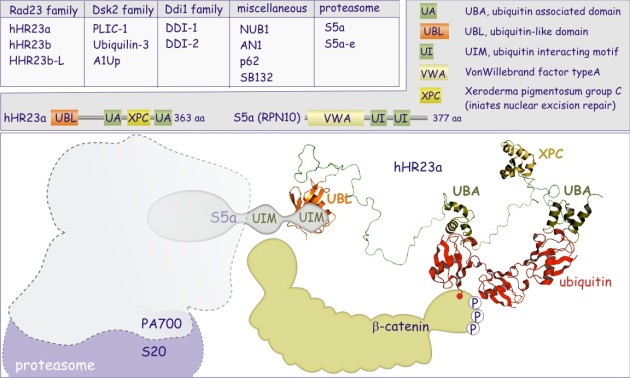
b/ le couvercle, composé de non-ATPases Rpn3-14 ). Rpt est l'acronyme de Regulatury Particle ATPase, Rpn est l'acronyme de Regulatory Particle Non-ATPase. Les différents rôles de Rpt's et Rpn's dans le fonctionnement du protéasome restent à préciser. On sait cependant que Rpt 5 joue le rôle de récepteur aux protéines poly-ubiquitinées. La présence de différentes ATPases démontre que la dégradation protéique est un processus dépendant de l'ATP : en effet, après désubiquitination, par le PA 700, la protéine est conduite à l'intérieur du cylindre où elle est découpée en peptides de 7 à 9 résidus. Rappelons que l'hydrolyse de la liaison peptidique n'est pas consommatrice d'ATP, étant elle-même un processus exergonique (qui libère de l'énergie).
La figure ci-dessous montre comment les protéines poly-ubiquitinées sont fixées par le complexe régulateur du protéasome.
![]() Pour en savoir plus : deux articles de revues sur le protéasome
Pour en savoir plus : deux articles de revues sur le protéasome
- Proteasomes machines for all reasons 2007-DeMartino (PDF) [pdf]
Complément : Excursion : l'immunoprotéasome et la présentation des antigènes viraux
Le protéasome joue aussi un rôle dans la régulation de la réponse immune à une infection virale. Pour se propager les virus piratent la machinerie de protéosynthèse de l'hôte pour produire des protéines virales qui seront assemblées en nouvelles particules virales qui quitteront les cellules pour en infecter d'autres. Dans le but d'alerter le système immunitaire, les cellules infectées doivent présenter en surface et dans le contexte du complexe majeur d'histocompatibilité classe I (CMH classe I), de petits peptides (appelés antigènes) issus de la coupure des protéines virales (c'est la présentation de l'antigène). Le complexe antigene-CMH classe I est reconnu par un récepteur porté par un lymphocyte T (T cell receptor, TCR) et ceci est le point de départ de la réponse immunoprotectrice.
La réponse immunitaire est rendue plus performante grâce à l'interféron–\(\gamma\) produit par l'organisme infesté. Plus précisément, l'interféron–\(\gamma\) favorise la présentation de l'antigène viral par son action sur la nature du protéasome. Chez les mammifères, l'interféron–\(\gamma\) induit l'expression de sous-unités spéciales, appelées pour cela « inductibles » (\(\rm \beta i1\), \(\rm \beta i2\) et \(\rm \beta i5\)). Il induit également l'expression d'une sous-unité spéciale constitutive de l'une des unités régulatrices, PA 700 (19 S), du protéasome. Ces deux modifications donnent naissance à un « immunoprotéasome » qui reconnaît sélectivement les substrats viraux et qui a des spécificités catalytiques différentes de celles du protéasome ordinaire. L'immunoprotéasome produit des peptides antigéniques d'environ 15 acides aminés qui se lient particulièrement bien avec le complexe CMH classe I et seront donc présentés en priorité à la surface des cellules infectées.