Processus d'ubiquitination
Cette voie de destruction implique l'étiquetage de la protéine à éliminer, par l'addition de molécules d'ubiquitine qui sera elle-même reconnue par un complexe protéolytique, le protéasome.
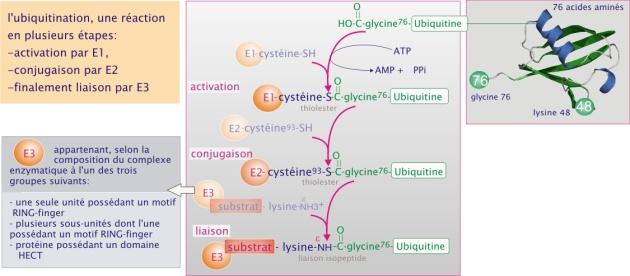
L'étiquetage consiste en la liaison sur la protéine à détruire de plusieurs unités d'ubiquitine (chaîne de 76 acides aminés, poids moléculaire 8,4 kDa). Ce système est utilisé aussi bien par les archae bactéries, que les levures, les plantes et les animaux. Ce processus d'ubiquitination est effectué par trois complexes enzymatiques différents : E1, ubiquitin-activating enzyme, E2, ubiquitin-conjugating enzyme et E3, ubiquitine-ligase. A ce jour chez l'Homme on a identifié un seul gène pour E1, plusieurs pour E2 et à peu près 300 pour E3. Dans la plupart des cas, E3 se comporte comme un récepteur, qui sélectionne et présente à l' E2, la protéine à détruire. Les nombreuses formes d' E3 ont chacune une affinité spécifique pour une gamme de protéines.
Remarque : Classification des ubiquitine ligases
E3 appartient, selon la composition du complexe enzymatique, à l'un des trois groupes suivants :
une seule unité possédant un motif RING-finger,
plusieurs sous-unités dont l'une possédant un motif RING-finger,
protéine possédant un domaine HECT.
La procédure d'ubiquitination est la suivante :
D'abord, en présence d'ATP l'ubiquitine réagit avec E1, formant ainsi un thiolester entre sa glycine C–terminale et un résidu cystéine d' E1 (voir figure 20). L'ubiquitine est ensuite transférée sur E2, réalisant une liaison instable avec une cystéine (dans la figure 20, c'est la cystéine-93). Enfin, l'ubiquitine est transférée sur une lysine du substrat par l' E3 (dans notre exemple de , l'E3 est un complexe multiprotéique). Le processus se répète en se servant de la lysine–48 de l'ubiquitine fixée sur le substrat, aboutissant à la formation d'une chaîne d'ubiquitines. Une chaîne d'au moins quatre molécules représente le signal de dégradation.
Remarque : La règle du N–terminal
La durée de la vie d'une protéine peut être déterminée par la composition de sa séquence N–terminale. Dans la levure, il a été montré que des protéines avec une méthionine N–terminale ont une demi-vie de 20 heures alors que celles qui ont une arginine dans cette position ne vivent que 2 minutes en moyenne. C'est ce qu'on appelle « la règle du N–terminal » (Alexander Varshavsky, 1996).
D'autres particularités de la séquence de la protéine déterminent aussi sa durée de vie. Par exemple, certaines formes d'E3 reconnaissent uniquement les protéines qui possèdent des acides animés phosphorylés formant des séquences précises telles que \(\rm -D-^*S-G-\phi -X-^*S- \) ou \(\rm -P-E-^*S-T-\)(appelées « »). Dans ce cas particulier le choix de la protéine à détruire est donc réalisé par une phosphorylation préalable effectuée par une enzyme appelée protéine kinase (exemple de , la glycogène synthase–3\(\beta\) (GSK–3\(\beta\)) qui a préalablement phosphorylé la \(\beta–\)caténine).
Prix Nobel pour la découverte du système d'ubiquitination
Pour plus d'information sur ces lauréates du Prix Nobel cliquez ici http://www.nobelprize.org/nobel_prizes/chemistry/laureates/2004/