5.2.2.2 - Distances évolutives
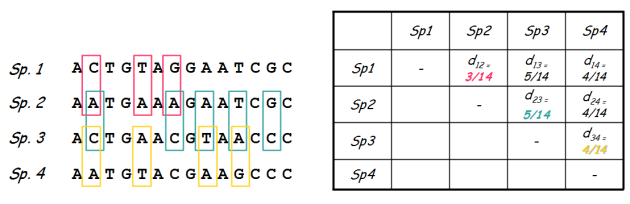
La ressemblance entre OTU peut être estimée par la quantification d'une mesure de distance. La ressemblance est alors décrite par une valeur numérique qui sera d'autant plus grande que la ressemblance est faible[1]. Les distances évolutives, notamment employées dans le cadre des phylogénies moléculaires, visent à quantifier le nombre de substitutions qui se sont accumulées sur chaque lignée évolutive depuis un ancêtre commun (voir chap 4.2.1). En vertu du principe de l'horloge moléculaire (voir chap 4.2.1), les distances évolutives peuvent être considérées à une constante près (la vitesse d'évolution) comme une approximation de la mesure du temps qui sépare deux OTU de leur ancêtre commun (fig. 5.33).
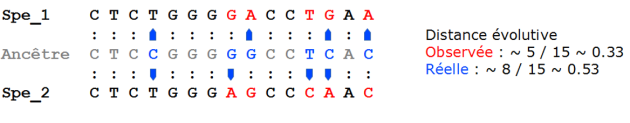
L'estimation du nombre de substitutions entre deux séquences depuis leur ancêtre commun se fait par la réalisation d'un alignement des séquences[2] et le dénombrement des substitutions entre ces séquences. Quand on compare plus de deux OTU, les distances évolutives (substitutions) sont estimées pour chaque couple d'OTU et les valeurs sont stockées dans une matrice de distances évolutives. Lorsque le temps de divergence est court, la distance évolutive entre deux OTU peut être estimée par le dénombrement des substitutions observées. Si le temps de divergence est plus long, le dénombrement des substitutions observées est le plus souvent une sous-estimation du nombre de substitutions réelles. Par exemple, en quatrième position de la séquence (fig. 5.34), la même substitution a eu lieu dans les deux lignées. La comparaison des deux séquences actuelles à cette position ne permet pas de repérer ces deux substitutions.