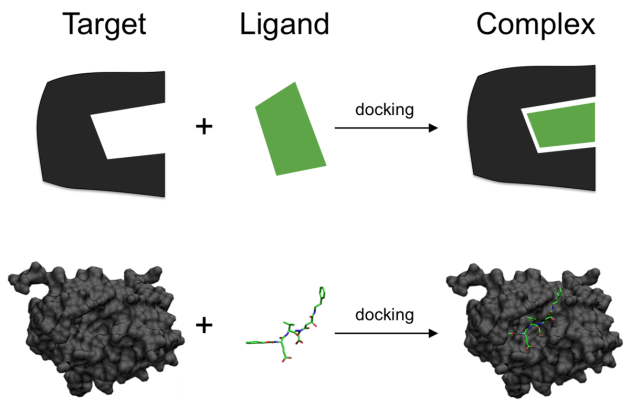
1. Comment les interactions moléculaires peuvent être utilisées pour prédire la fonction d'un candidat pharmacologiquement actif ?
Dans le monde du vivant, les activités biologiques sont majoritairement liées à la réponse des récepteurs biologiques suite à un stimulus chimique. Un partie significative de la biologie est basée sur un paradigme suggérant que la séquence peptidique ou d'acides-aminés dicte la structure, qui dicte elle-même la fonction des récepteurs.
Dès lors, il s'agit de pouvoir prédire les interactions entre stimulus et les récepteurs biologiques sur la base de leur structure.
Le drug design va donc tenter d'utiliser les informations sur les interactions entre molécules et récepteurs pour optimiser l'usage de ces molécules candidates.
Exemple : Relenza
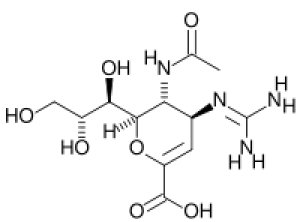
Le Relenza est l'une des premières molécules conçues par drug design. Elle visait la lutte contre le virus influenza, en ciblant l'inhibition de la neuramidase. Les chercheurs ont conçu cette molécules en optimisant son interaction avec l'enzyme, pour bloquer son action. Le critère de sélection était l'optimisation de l'énergie d'interaction entre les deux partenaires. Le relenza est donc un inhibiteur compétitif, il bloque le site actif de la neuraminidase et empêche le développement de l'infection.
Les virus grippaux Influenza A sont nommés sous la forme HxNy (H1N1, etc), avec x le type d'hémagglutinine et y le type de neuraminidase. Il en existe 144 (16 types d'hemaglutine × 9 types de neuraminidase).