Terminaison de la traduction
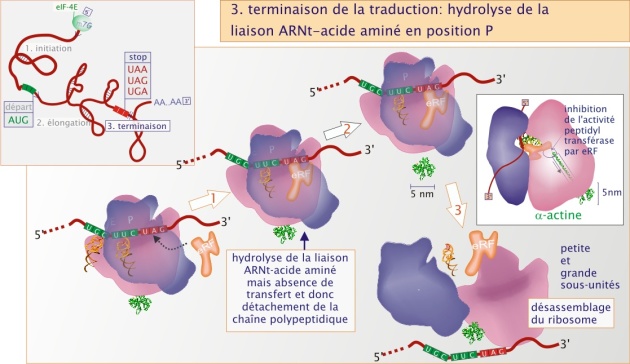
La translocation s'effectue jusqu'à l'un des codons stop, UAA, UAG ou UGA, qui est lié à une protéine appelée facteur de terminaison de l'élongation (eTF, également connu sous le nom de eukaryotic Release Factor (eRF)), elle-même associée au GTP. Aucun autre acide aminé ne peut plus être ajouté, et la traduction est terminée par l'hydrolyse de la liaison du dernier acide aminé avec son ARNt (c'est ARNtphe dans la figure 15, ), formant ainsi l'extrémité carboxy terminale de la protéine. La chaîne protéique alors complète est libérée et les deux sous-unités du ribosome se dissocient libérant ainsi l'ARNm, .
![]() Voir une version 3D animée de la synthèse des protéïnes.
Voir une version 3D animée de la synthèse des protéïnes.
Macromedia Flash - 3,3Mo
Venkatraman Ramakrishnan, Thomas Steitz and Ada Yonath, ont été recompensés en 2009 par le Prix Nobel de Chimie, "for studies of the structure and function of the ribosome".
Pour en savoir plus, cliquez sur ce lien http://nobelprize.org/nobel_prizes/chemistry/laureates/2009/.
Pour plus d'information sur la structure et fonction du ribosome procaryote, nous suggérons le site web du laboratoire de Dr V. Ramakrishnan (MRC, Cambridge, Royaume Uni), cliquez sur ce lien http://www.mrc-lmb.cam.ac.uk/ribo/homepage/.
![]() Pour en savoir plus, cliquez sur ce lien "review on ribosomes 2002-ramakrishnan [pdf]"
Pour en savoir plus, cliquez sur ce lien "review on ribosomes 2002-ramakrishnan [pdf]"
Complément : Quelques données concrètes
Pour un fibroblaste humain, la vitesse de traduction d'un ARNm est estimée à 5 - 10 acides aminés par seconde, autrement dit une protéine de 50 kDa (poids moléculaire courant) est synthétisée en environ 64 secondes. Pendant ce temps, environ 490 acides aminés sont liés, nécessitant l'hydrolyse de 980 ATP (convertis en ADP) et 980 GTP (convertis en GDP). Détail de cette évaluation (« n » étant le nombre d'acides aminés liés) : 2n ATP utilisés pour le chargement des ARNt en acides aminés, 1 GTP pour l'initiation de traduction (eIF2), n-1 GTP pour le positionnement des ARNt et la réalisation de n-1 liaisons peptidiques, n-1 GTP pour la translocation ribosome/ARNm et un GTP pour la terminaison de la traduction.
Grâce au « proofreading », les erreurs de traduction sont rares : une erreur pour 10 000 acides aminés, donc environ une protéine de 50 kDa sur 20 contient un acide aminé non correct. Les sources d'erreurs sont : attachement d'un acide aminé non conforme à l'ARNt et appariement erroné entre le codon et anticodon. L'erreur qui changerait profondément la conformation de la protéine conduirait à son élimination par le protéasome (voir chapitre 07, la dégradation des protéines).